7일차(지도학습과 비지도학습)
##와인 데이터 가져오기
import pandas as pd
wine=pd.read_csv("https://bit.ly/wine-date")
wine
##문제지와 정답지 나누기
data=wine[['alcohol','sugar','pH']].to_numpy()
target=wine['class'].to_numpy()
##일정한 비율로 데이터 나누기
from sklearn.model_selection import train_test_split
train_input,test_input,train_target,test_target=train_test_split(data,target,test_size=0.2)
##데이터 전처리하기
from sklearn.preprocessing import StandardScaler
ss=StandardScaler()
ss.fit(train_input)
train_scaled=ss.transform(train_input)
test_scaled=ss.transform(test_input)
##로지스틱 라이브러리를 통해 학습시키고 성능 알아보기
>from sklearn.linear_model import LogisticRegression
>lr=LogisticRegression()
>lr.fit(train_scaled,train_target)
>
>print(lr.score(train_scaled,train_target))
>print(lr.score(test_scaled,test_target))
0.7804502597652492
0.7838461538461539
##결정트리 라이브러리를 통해 학습시키고 성능 알아보기
>from sklearn.tree import DecisionTreeClassifier
>dt=DecisionTreeClassifier()
>dt.fit(train_scaled,train_target)
>
>print(dt.score(train_scaled,train_target))
>print(dt.score(test_scaled,test_target))
0.9978833942659227
0.8653846153846154
##결정 트리 그리기
from sklearn.tree import plot_tree #아준 완벽하게 나눈것...와인을,,,,과대적합..
import matplotlib.pyplot as plt
plt.figure(figsize=(10,10))
plot_tree(dt)
plt.show()

##결정트리의 깊이를 3으로 설정하고 학습시킨 후, 성능 알아보기
>dt=DecisionTreeClassifier(max_depth=3)
>dt.fit(train_input,train_target)
>
>print(dt.score(train_input,train_target))
>print(dt.score(test_input,test_target))
0.8454877814123533
0.8515384615384616>>과대 적합을 막은 것을 확인할 수 있다.
##다시 학습시킨 후의 결정 트리 그리기
plt.figure(figsize=(20,20))
plot_tree(dt,filled=True,feature_names=['alcohol','sugar','pH'])
plt.show()>>filled=색깔의 여부(화이트 와인/레드 와인)

##요인의 중요도 보기
>dt.feature_importances_
array([0.12476471, 0.8701314 , 0.0051039 ])>>당도가 가장 중요하다는 것을 알 수 있다.
>>gini 는 불순도를 나타낸다.
##이제까지 훈련시키고 테스트했던 방법은
1.훈련데이터로 학습하고, 훈련데이터 평가해서 100점
2.훈련데이터랑 시험데이터를 나누어 훈련 시키고 테스트 하기
>>이런 방법들은 문제점이 있다.-->>과소,과대 적합 확인해서 이를 막을 수 있게 반복적으로 고치는 것은 결국 시험 데이터에 맞추는 행동이다. 이런 식으로 하면 또 새로운 데이터가 들어왔을 때, 과대 적합,과소 적합이 될 수 있다.
맞추는 행동을 해서는 안된다.
그럼, 과소, 과대 적합을 어떻게 막아야 할까??
최대한 훈련 데이터 안에서 막아야 한다. 중간 중간에 쪽지 시험 봐가며 과대,과소 적합을 테스트하고
후에 시험 데이터로 평가해야 한다.그래도 과소 과대 적합이 나온다면, 그건 어쩔 수 없는 것이다.
#쪽지시험 : 검증데이터
##80%: 훈련 데이터 ,20%: 시험 데이터
##훈련 데이터 중 80%는 sub 데이터, 20% 는 val 데이터(검증 데이터)
##훈련 데이터 한번 더 나누어 검증 데이터 뽑아내기
sub_input,val_input,sub_target,val_target=train_test_split(train_input,train_target,test_size=0.2)
##결정트리를 이용하여 sub 데이터 학습시키고 sub 데이터와 val 데이터 성능 알아보기
>dt=DecisionTreeClassifier()
>dt.fit(sub_input,sub_target) #val=검증데이터,
>print(dt.score(sub_input,sub_target))
>print(dt.score(val_input,val_target))
0.997834977146981
0.8538461538461538>>과대 적합 된 것을 확인 할 수 있다.
##교차 검증 라이브러리를 이용하여 학습시키기
>from sklearn.model_selection import cross_validate #교차검증 라이브러리
>
>scores=cross_validate(dt,train_input,train_target)
>scores
{'fit_time': array([0.00767374, 0.00753045, 0.00746036, 0.0082593 , 0.00781035]),
'score_time': array([0.00092244, 0.00082827, 0.00089645, 0.00116634, 0.00092411]),
'test_score': array([0.88461538, 0.85 , 0.86333013, 0.86333013, 0.83830606])}>>5 번 학습시킨 것을 확인 할 수 있다.
##5번의 쪽지 시험 점수 평균내기
>import numpy as np
>np.mean(scores['test_score'])
0.8599163396757236
##랜덤 포레스트를 이용하여 성능 알아보기
from sklearn.ensemble import RandomForestClassifier
rf=RandomForestClassifier()
scores=cross_validate(rf,train_input,train_target,return_train_score=True)>>랜덤 포레스트란 결정 트리를 여러 개 만들어서 가장 좋은 결정 트리를 고르는 것이다.
##훈련 데이터와 검증 데이터의 성능 평균 알아보기
>print(np.mean(scores['train_score']),np.mean(scores['test_score']))
0.9982201362283153 0.8837776708373435>>과대 적합은 됐지만 검증 데이터의 성능이 더 좋아진 것을 확인 할 수있다.
##랜덤 포래스트로 훈련 데이터 학습시키기
>rf=RandomForestClassifier(oob_score=True)
>rf.fit(train_input,train_target)
RandomForestClassifier(bootstrap=True, ccp_alpha=0.0, class_weight=None,
criterion='gini', max_depth=None, max_features='auto',
max_leaf_nodes=None, max_samples=None,
min_impurity_decrease=0.0, min_impurity_split=None,
min_samples_leaf=1, min_samples_split=2,
min_weight_fraction_leaf=0.0, n_estimators=100,
n_jobs=None, oob_score=True, random_state=None,
verbose=0, warm_start=False)
##요인의 중요도 알아보기
>rf.feature_importances_
array([0.23768398, 0.49096339, 0.27135264])>>당도가 가장 중요하다고 본다.
##검증 데이터의 성능 알아보기
>rf.oob_score_
0.8939772945930344>>남는 데이터로 중간 중간 쪽지시험을 친 점수이다.
>>따로 시험데이터가 필요하지 않다.
##히스토그램 기반 그레이디언트 부스팅 이용하여 교차검증 학습시키기
from sklearn.experimental import enable_hist_gradient_boosting
from sklearn.ensemble import HistGradientBoostingClassifier
hgb=HistGradientBoostingClassifier()
scores=cross_validate(hgb,train_input,train_target,return_train_score=True)
##훈련데이터와 시험 데이터 성능의 평균 알아보기
>print(np.mean(scores['train_score']),np.mean(scores['test_score']))
0.9303924498776557 0.8731927889242614>>과대 적합 조금 막아준 것을 확인 할 수 있다.
----------
지금까지는 지도 학습을 해보았다.
##지도 학습:문제지와 정답지를 주고 컴퓨터가 학습하게 하는 것
##비지도 학습 : 문제지만 주는 것
##비지도 학습은 언제 사용할까??패턴을 찾아서 라벨링하기 위해서
픽셀값의 평균을 이용한다.
ex) 강아지 픽셀값 평균=37
고양이 픽셀값 평균=115
새로운 데이터 픽셀값=117 이라면 고양이 라고 결정한다.
#비지도 학습
##데이터 저장하기
!wget https://bit.ly/fruits_300_data -O fruits_300.npy
fruits = np.load('fruits_300.npy')
fruits>>#0에 가까울 수록 흰색 255에 가까울수록 검은색
##데이터 형태 알아보기
>fruits.shape
(300, 100, 100)>>사진이 300장이 있고 가로 100, 세로 100을 의미한다.
##과일 데이터의 첫번째 사진 보기
plt.imshow(fruits[0],cmap='gray_r')
plt.show()
##사진 2개 그려보기
fig,axs=plt.subplots(1,2) #1행 2열(즉, 자리를 2개 할당하는 것)
axs[0].imshow(fruits[100],cmap='gray_r')
axs[1].imshow(fruits[200],cmap='gray_r')
plt.show()
>>파인애플과 바나나 사진을 볼 수 있다.
>>0~99 : 사과, 100~199: 파인애플, 200~299: 바나나
##데이터를 컴퓨터가 인식하기 좋은 형태로 바꾸기
>fruits2=fruits.reshape(-1,10000)
>fruits2.shape
(300, 10000)>>사진 데이터를 300장이며 일자형 데이터로 바꾸어 주었다.
##데이터 출력하기
>fruits2
array([[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1],
...,
[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1]], dtype=uint8)
##사과, 파인애플, 바나나 데이터 나누기
apple=fruits2[:100]
pineapple=fruits2[100:200]
banana=fruits2[200:]
##사과 데이터 형태 알아보기
>apple.shape
(100, 10000)>>100장에 10000개의 픽셀을 가지고 있는 데이터이다.
##
>apple
array([[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1],
...,
[1, 1, 1, ..., 1, 1, 1],
[2, 2, 2, ..., 1, 1, 1],
[1, 1, 1, ..., 1, 1, 1]], dtype=uint8)>>가로 한 줄의 수 개수=100000개
>>가로로 평균을 내면 사진을 평균 내는 것 :100개의 평균 값이 나온다.
>>세로로 평균을 내면 픽셀을 평균 내는 것 : 100000개의 평균 값이 나온다.
##데이터 세로로 평균 내기
>apple.mean(axis=0)
array([1.01, 1.01, 1.01, ..., 1. , 1. , 1. ])>>axis=1 :가로로 평균, axis=0 : 세로로 평균 내는 것을 의미한다.
>>10000개의 데이터가 나온다.
##데이터 가로로 평균 내기
>apple.mean(axis=1)
array([ 88.3346, 97.9249, 87.3709, 98.3703, 92.8705, 82.6439,
94.4244, 95.5999, 90.681 , 81.6226, 87.0578, 95.0745,
93.8416, 87.017 , 97.5078, 87.2019, 88.9827, 100.9158,
92.7823, 100.9184, 104.9854, 88.674 , 99.5643, 97.2495,
94.1179, 92.1935, 95.1671, 93.3322, 102.8967, 94.6695,
90.5285, 89.0744, 97.7641, 97.2938, 100.7564, 90.5236,
100.2542, 85.8452, 96.4615, 97.1492, 90.711 , 102.3193,
87.1629, 89.8751, 86.7327, 86.3991, 95.2865, 89.1709,
96.8163, 91.6604, 96.1065, 99.6829, 94.9718, 87.4812,
89.2596, 89.5268, 93.799 , 97.3983, 87.151 , 97.825 ,
103.22 , 94.4239, 83.6657, 83.5159, 102.8453, 87.0379,
91.2742, 100.4848, 93.8388, 90.8568, 97.4616, 97.5022,
82.446 , 87.1789, 96.9206, 90.3135, 90.565 , 97.6538,
98.0919, 93.6252, 87.3867, 84.7073, 89.1135, 86.7646,
88.7301, 86.643 , 96.7323, 97.2604, 81.9424, 87.1687,
97.2066, 83.4712, 95.9781, 91.8096, 98.4086, 100.7823,
101.556 , 100.7027, 91.6098, 88.8976])
>>100개의 데이터가 나온다.
##각 사진들의 평균 값들 히스토그램으로 나타내기
plt.hist(np.mean(apple,axis=1),alpha=0.5)
plt.hist(np.mean(pineapple,axis=1),alpha=0.5)
plt.hist(np.mean(banana,axis=1),alpha=0.5)
plt.legend(['apple','pineapple','banana'])
plt.show()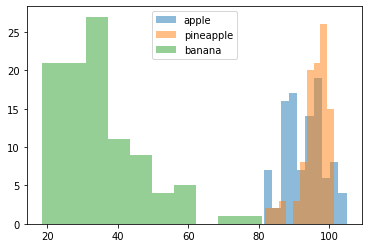
>>사과와 파인애플이 겹치는 부분이 많은 것으로 보아, 두 데이터를 구분하는 것이 어려울 것으로 보인다.
##사과, 파인애플, 바나나의 픽셀 평균값을 그래프로 나타내기
fig,axs=plt.subplots(1,3,figsize=(20,5)) #v픽셀 평균넨값
axs[0].bar(range(10000),np.mean(apple,axis=0))
axs[1].bar(range(10000),np.mean(pineapple,axis=0))
axs[2].bar(range(10000),np.mean(banana,axis=0))
plt.show()
##각 데이터 별로 평균 낸 값을 다시 사진 형태로 만들어주고 그래프로 나타내기
apple_mean=np.mean(apple,axis=0).reshape(100,100) # 사과 사진의 픽셀값들을 평균내서 그것을 다시 사진으로 바꾼다.
pineapple_mean=np.mean(pineapple,axis=0).reshape(100,100)
banana_mean=np.mean(banana,axis=0).reshape(100,100)
fig,axs=plt.subplots(1,3,figsize=(20,5))
axs[0].imshow(apple_mean,cmap='gray_r')
axs[1].imshow(pineapple_mean,cmap='gray_r')
axs[2].imshow(banana_mean,cmap='gray_r')
plt.show()
>>사진들의 픽셀값을 평균낸 값을 다시 사진으로 바꾸었을 때의 그림이다.
##비지도 학습의 원리
>>과일 사진 300장-사과의 평균 값 사진=사과의 평균값이 빠진 과일 사진--> 픽셀이 다 깨졌을 것..
>>작은 값들 순으로 오름차순,작은 값부터 100장은 사과사진일 것이다.
##과일사진-사과사진 값 절대값으로 출력하기
#과일-사과의 편균=사과의 편군값이 빠진 과일 사진
#작은 값들 순으로 오름차순 작은 값부터 100장은 사과 사진
#0에 가까우면 그사진이네...
>abs_diff=np.abs(fruits-apple_mean)
>abs_diff
array([[[0.01, 0.01, 0.01, ..., 0. , 0. , 0. ],
[0.01, 0.01, 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
...,
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ]],
..................생략....................
[[0.01, 0.01, 0.01, ..., 0. , 0. , 0. ],
[0.01, 0.01, 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
...,
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ],
[0. , 0. , 0. , ..., 0. , 0. , 0. ]]])
##사진 형태 알알보기
>abs_diff.shape
(300, 100, 100)
##과일 사진-사과 사진한 300장 모두 평균내기
>abs_mean=np.mean(abs_diff,axis=(1,2))
>abs_mean
array([17.37576 , 13.508874, 17.183394, 15.68311 , 17.983306, 20.565392,
16.795812, 16.144276, 19.611994, 21.32059 , 16.454222, 16.590134,
13.37039 , 17.23263 , 15.92806 , 15.496638, 18.582212, 16.48196 ,
27.651556, 19.7871 , 20.826912, 16.417934, 17.059946, 15.904296,
15.086176, 19.371364, 24.076362, 14.777732, 19.20517 , 20.805182,
.............................생략................................
79.509962, 74.302932, 64.071044, 69.071934, 71.144022, 74.172134,
73.076824, 79.077932, 65.437208, 68.371726, 73.046472, 71.420232,
71.167158, 64.457272, 79.541768, 67.113442, 65.346244, 71.26783 ,
71.757494, 71.05042 , 76.028654, 70.09248 , 72.439504, 73.240238])
##오름 차순으로 정렬된 데이터 인덱스 확인하기
>apple_index=np.argsort(abs_mean)
>apple_index
array([ 33, 48, 70, 57, 87, 12, 78, 59, 1, 74, 86, 38, 50,
92, 69, 27, 68, 30, 66, 24, 76, 98, 15, 84, 47, 90,
3, 94, 53, 23, 14, 71, 32, 7, 73, 36, 55, 77, 21,
............................생략................................
294, 220, 261, 202, 208, 243, 229, 298, 240, 269, 271, 286, 282,
233, 299, 239, 235, 281, 211, 256, 277, 268, 248, 254, 247, 201,
270, 265, 296, 215, 249, 255, 252, 258, 219, 266, 238, 200, 283,
253, 264, 276, 290, 210, 263, 203, 244, 241, 207, 272, 267, 250,
225])
##비지도 학습으로 사과 사진만 뽑아내기
fig,axs=plt.subplots(10,10,figsize=(10,10))
for i in range(10):
for j in range(10):
axs[i,j].imshow(fruits[apple_index[i*10+j]],cmap='gray_r')
axs[i,j].axis('off')
plt.show()
>>비지도 학습으로 사과 사진만 뽑아낸 것을 확인 할 수 있다.
##비지도 학습으로 파인애플 사진만 뽑아내기
abs_diff=np.abs(fruits-pineapple_mean)
abs_mean=np.mean(abs_diff,axis=(1,2))
apple_index=np.argsort(abs_mean)
fig,axs=plt.subplots(10,10,figsize=(10,10)) #비지도 학습으로 사과사진만 뽑아냄
for i in range(10):
for j in range(10):
axs[i,j].imshow(fruits[apple_index[i*10+j]],cmap='gray_r')
axs[i,j].axis('off')
plt.show()

>>사과가 3개 껴있지만 다수 파인애플을 뽑아내며 비지도 학습이 잘 되었다고 볼 수 있다.
##비지도 학습으로 바나나 사진만 뽑아내기
abs_diff=np.abs(fruits-banana_mean)
abs_mean=np.mean(abs_diff,axis=(1,2))
apple_index=np.argsort(abs_mean)
fig,axs=plt.subplots(10,10,figsize=(10,10)) #비지도 학습으로 사과사진만 뽑아냄
for i in range(10):
for j in range(10):
axs[i,j].imshow(fruits[apple_index[i*10+j]],cmap='gray_r')
axs[i,j].axis('off')
plt.show()
#여기서 문제점!!비지도 학습 하는 이유는 라벨링이다.
우리는 이미 무슨 사진이 있는지 알고 있었고 3가지의 과일 사진으로 나눠야한다는 것을 알고 있는 상황이다. 답을 알고
원래 비지도 학습은 몇개의 데이터로 라벨링하는지 모르는 상태에서 학습이 이루어진다.
#이번에는 데에터에 뭐가 있는지 모를때를 해보겠다.
##라이브러리를 통해 비지도 학습시키기
>from sklearn.cluster import KMeans
>
>km=KMeans(n_clusters=3)
>km.fit(fruits2)
KMeans(algorithm='auto', copy_x=True, init='k-means++', max_iter=300,
n_clusters=3, n_init=10, n_jobs=None, precompute_distances='auto',
random_state=None, tol=0.0001, verbose=0)
##라벨링 조회하기
>km.labels_
array([1, 1, 1, 1, 1, 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 1,
2, 1, 1, 1, 1, 1, 1, 1, 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 1, 1,
1, 1, 1, 1, 1, 1, 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 2, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], dtype=int32)
##라벨링한 데이터 별로 개수 세기
>np.unique(km.labels_,return_counts=True)
(array([0, 1, 2], dtype=int32), array([ 98, 91, 111]))
##사진 개수에 따라 그림 그려주는 함수 만들기
def draw_fruits(arr,ratio=1):
n=len(arr) #사진 몇게 있는지 판단
rows=int(np.ceil(n/10)) #ceil올림함수//몇행으로 결정
cols=n if rows<2 else 10 #만약에 1행 열을 10으로 고정
fig,axs=plt.subplots(rows,cols,figsize=(cols*ratio,rows*ratio),squeeze=False)
for i in range(rows):
for j in range(cols):
if i*10+j<n:
axs[i,j].imshow(arr[i*10+j],cmap='gray_r')
axs[i,j].axis('off')
plt.show()
##함수를 이용하여 0으로 라벨링 된 데이터 출력하기
draw_fruits(fruits[km.labels_==0])
>>0은 바나나였다.
#지금 까지는 과일이 몇 종류가 있는지 알려주고 학습 시켰다면, 이제 몇 종류의 과일이 있는 지 모르는 상태에서 학습시키고 성능 알아보기
##군집 개수에 따른 inertia 구하기
inertia=[]
for k in range(2,7):
km=KMeans(n_clusters=k)
km.fit(fruits2)
inertia.append(km.inertia_)
plt.plot(range(2,7),inertia)
plt.show()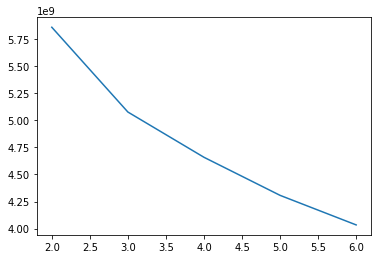
>>드라마틱하게 떨어지는 구간이 최적의 군집 개수 알 수 있다.
>>3개가 최적은 군집 개수
----비지도 학습 끝------
##주성분 분석=PCA(principal component analysis) : 차원 축소 기법으로 군집이나 분류에 영항을 끼치지 않고 데이터의 용량을 줄이는 방법
##데이터 형태 보기
>fruits2.shape
(300, 10000)
##주성분 분석 라이브러리를 이용하여 픽셀 줄이기
>from sklearn.decomposition import PCA
>pca=PCA(n_components=50) #50개의 주성분을 찾을거다.
>pca.fit(fruits2) #fruits로 찾아라!1-50위
PCA(copy=True, iterated_power='auto', n_components=50, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
>>주성분을 50개 찾는 것을 의미한다.
##데이터 형태 보기
>pca.components_.shape
(50, 10000)>>50개의 주성분이 있고, 각각의 주성분은 10000개의 데이터를 기반으로 한다.
##사진 그림 그려보기
draw_fruits(pca.components_.reshape(-1,100,100))
>>데이터 형태를 이미지로 바꾸어 확인했다.
>>특징을 잡아낸 것을 볼 수 있다.
##데이터 형태 보기
>fruits2.shape
(300, 10000)
>>사진 300개에 픽셀이 10000개
##데이터 줄이기
>fruits_pca=pca.transform(fruits2)
>fruits_pca.shape
(300, 50)>>10000개의 픽셀을 50개로 줄인 것을 확인 할 수 있다.
##다시 픽셀 늘리기
>fruits_inverse=pca.inverse_transform(fruits_pca)
>fruits_inverse.shape
(300, 10000)>>50개의 픽셀을 다시 10000개로 늘리기
##데이터 사진 형태로 바꾸고 사진 출력하기
fruits_reconstruct=fruits_inverse.reshape(-1,100,100)
for i in [0,100,200]:
draw_fruits(fruits_reconstruct[i:i+100])
print('\n')


>>잘 보관되었다.
>>학습해도 문제없이 구분 할 것으로 예상된다.
##컴퓨터가 픽셀을 50개로 줄였을때 이해할수 있는 %보기
>np.sum(pca.explained_variance_ratio_)
0.9215066896668017>>픽셀을 줄여도 원본은 92%로 이해한다는 것을 의미한다.
##정답지 만들기
>target=np.array([0]*100+[1]*100+[2]*100)
>target
array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2])
##픽셀을 10000개로 했을 때의 정확도 보기
>from sklearn.linear_model import LogisticRegression
>from sklearn.model_selection import cross_validate
>lr=LogisticRegression()
>
#픽셀 10000개로 했을 때 정확도 : 99.6%
>scores=cross_validate(lr,fruits2,target)
>print(np.mean(scores['test_score']))
0.9966666666666667
##픽셀을 50개로 했을 때의 정확도 보기
>scores=cross_validate(lr,fruits_pca,target) #픽셀 50개로 했을때, 정확도 100%
>print(np.mean(scores['test_score']))
1.0
##주성분 요인 2개로 데이터 줄이기
>pca=PCA(n_components=2)
>pca.fit(fruits2)
PCA(copy=True, iterated_power='auto', n_components=2, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
##설명력 보기
>np.sum(pca.explained_variance_ratio_)
0.5229877245800597
##주성분 개수 확인하기
>fruits_pca=pca.transform(fruits2)
>fruits_pca.shape
(300, 2)
##정확도 확인하기
>scores=cross_validate(lr,fruits_pca,target) #픽셀 2개로 했을때, 정확도 100%
>print(np.mean(scores['test_score']))
0.9933333333333334
##픽셀 2개로 학습시키기
#픽셀 2개짜리
>km=KMeans(n_clusters=3)
>km.fit(fruits_pca)
KMeans(algorithm='auto', copy_x=True, init='k-means++', max_iter=300,
n_clusters=3, n_init=10, n_jobs=None, precompute_distances='auto',
random_state=None, tol=0.0001, verbose=0)
##라벨링 된 값의 수 알아보기
>np.unique(km.labels_,return_counts=True)
(array([0, 1, 2], dtype=int32), array([ 91, 99, 110]))
##라벨링한 그림 그리기
for i in range(0,3):
draw_fruits(fruits[km.labels_==i])
print('\n')


>>라벨링이 잘 된것을 확인 할 수 있다.
##라벨링된 데이터 별로 산점도 그리기
#pca=PCA(n_components=0.9) #설명력의 수% 만큼 찾아라
#pca.fit(fruits2)
for i in range(0,3):
data=fruits_pca[km.labels_==i]
plt.scatter(data[:,0],data[:,1])
plt.show()